Akwizycja i przetwarzanie obrazu
Do badania włączono 52 pacjentów z NPC otrzymujących radioterapię w Fujian Cancer Hospital w latach 2020-2021. Badanie to zostało zatwierdzone przez Komisję Etyki Fujian Cancer Hospital (nr etyki: SQ2020-043-01), a wszyscy pacjenci wyrazili pisemną świadomą zgodę przed włączeniem do badania. Wszystkie metody wykonano zgodnie z Deklaracją Helsińską oraz odpowiednimi wytycznymi i przepisami. Podczas symulacji planowania leczenia obrazy CT zostały uzyskane na aparacie Brilliance CT Big Bore (Philips Medical Systems Inc., Cleveland, OH, USA) z protokołem głowa-szyja (120 kV, 225 mA). Wycinek tomogramu ma wymiary 512 x 512 pikseli, a rozdzielczość woksela to 1,14 x 1,14 x 3 mm.3. Wszystkie obrazy CBCT wykonano przed pierwszą radioterapią pacjentów w dniu XVI dla akceleratora Elekta Axesse, z napięciem lampy 120 kV i prądem ekspozycji 25 mA. Wymiary wycinka obrazu CBCT wynosiły 410 x 410 pikseli przy rozdzielczości 1 x 1 x 1 mm3.
Obrazy CT i CBCT rejestrowano ściśle według standardu dla obrazów CBCT, używając oprogramowania open source 3D-Slicer.25. Obrazy tomografii osiowej dopasowane do wokseli CBCT i obrazów objętościowych, zwanych RCT, zostały następnie ponownie próbkowane jako wzorzec odniesienia do oceny obrazu. Maski binarne zostały stworzone w oparciu o segmentację progową i metody przetwarzania morfologicznego, aby uniknąć szkodliwego wpływu struktur nieanatomicznych podczas procesu szkolenia. Wartości wokseli obrazów zostały przycięte do zakresu [− 1000, 2000]podczas gdy wartości wokseli regionów poza maskami zostały ustawione na −1000 HU.
Przed treningiem modelu CycleGAN wszystkie obrazy RCT i CBCT zostały przycięte od środka obrazu do rozmiaru 256 × 256, a wartość CT znormalizowano do [− 1, 1]. Do zestawu szkoleniowego wybrano losowo 41 pacjentów, a 11 pacjentów wykorzystano do walidacji. Z zestawu danych każdego pacjenta pobrano 264 wycinki. Zestaw danych szkoleniowych i walidacyjnych składał się zatem odpowiednio z 10 824 i 2 904 wycinków CT i CBCT. Ze względu na ograniczenia pamięci GPU, w tym badaniu przyjęto model 2D CycleGAN.
Kalibracja HU przez Phantom
Model CIRS 062 (CIRS Tissue Simulation Technology, Norfolk, VA, USA) został zeskanowany przy użyciu tego samego Big Bore CT i tego samego CBCT na akceleratorze liniowym, z tymi samymi parametrami akwizycji. Dla każdego skanu średnia liczba HU każdego materiału wejściowego (gęstość elektronów w stosunku do wody 1,00, 0,20, 0,50, 0,97, 0,99, 1,06, 1,07, 1,16 i 1,61) została odczytana w centralnym wycinku fantomu. Średnią liczbę HU w skanie CT i skanie mikro-CT wykreślono następnie odpowiednio w stosunku do znanej gęstości elektronów. HU zostało skorygowane na podstawie obrazów CBCT opartych na tych dwóch krzywych za pomocą wewnętrznego oprogramowania do tworzenia poprawionych obrazów CBCT (CBCT_cor).
Metoda CycleGAN
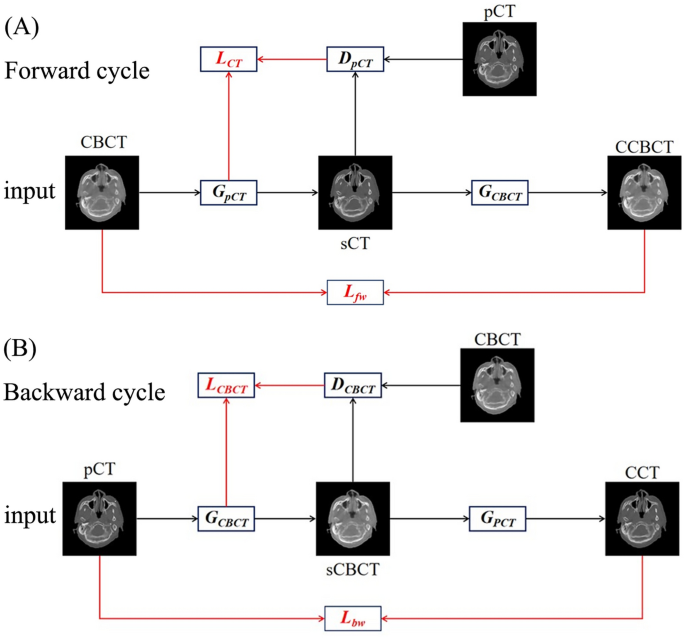
Jak pokazano na rysunku 1, model CycleGAN obejmuje dwa generatory i dwa dyskryminatory. W cyklu do przodu Generator-RCT (GRCT) generuje sCT z CBCT, a następnie Generator-CBCT (GCBCT) generuje cykl CBCT (CCBCT) z sCT. Będąc w cyklu wstecznym, użyj GCBCT Złożona CBCT (sCBCT) generuje z RCT, a następnie GRCT Generuje cykl CT (CCT) z sCBCT. Wytrawny, DRCT przyjaźńCBCT, aby określić, czy sCT i sCBCT są prawdziwymi obrazami. Funkcja utraty CycleGAN składała się z utraty antagonizmu i utraty spójności cyklu. Antagonistyczne straty dla dwóch cykli są
$$L_{CT} = E_{RCT}\lewo[ {\left( {1 – D_{RCT} (RCT)} \right)^{2} } \right] +E_{CBCT}\lewo[ {\left( {D_{RCT} \left( {G_{RCT} (CBCT)} \right)} \right)^{2} } \right] $$
(1)
I
$$ L_{CBCT} = E_{CBCT} \ lewo[ {\left( {1 – D_{CBCT} (CBCT)} \right)^{2} } \right] +E_{RCT}\lewo[ {\left( {D_{CBCT} \left( {G_{CBCT} (RCT)} \right)} \right)^{2} } \right] $$
(2)
Ilustracja generatywnej sieci przeciwstawnej dopasowanej do cyklu (CycleGAN).
Straty spójności sesji dla dwóch sesji wynoszą
$$ L_{fw} = E_{CBCT}\lewo[ {\left\| {CBCT – G_{CBCT} (G_{RCT} (CBCT))} \right\|_{1} } \right] $$
(3)
I
$$L_{bw}=E_{RCT}\lewo[ {\left\| {RCT – G_{RCT} \left( {G_{CBCT} (RCT)} \right)} \right\|_{1} } \right] $$
(4)
Tak więc, łącząc te dwa rodzaje strat, chodzi o to, aby:
$$ L_{cyklgan} = L_{CT} + L_{CBCT} + \lambda \left ( {L_{fw} + L_{bw}} \right) $$
(5)
Struktura i parametry sieci
Generator zawiera warstwę kodera, warstwę konwersji i warstwę dekodera. Koder redukuje liczbę wymiarów przestrzennych i definiuje cechy obrazu wejściowego. Warstwa transformacji, na którą składa się dziewięć warstw modułu ResNet26, zmienią się następnie na ich wektory własne. Dekoder ustala wymiary przestrzenne obiektu i generuje złożony obraz. Dyskryminator jest siecią binarną z wyjściami pomiędzy [0, 1]. Tryb jest trenowany za pomocą optymalizatora Adam27 z tensorflow28. Szybkość uczenia spada liniowo po 20 epokach z początkową wartością 0,0002, podczas gdy człon pędu β1 i 2 są ustawione na 0,5. Inne parametry są ustawione w następujący sposób: λ = 10, wielkość partii = 2, epoka = 100. Oryginalne obrazy CBCT i obrazy CBCT_cor są używane odpowiednio do uczenia modelu. W poniższym tekście SCT1 jest generowany przez model CycleGAN z oryginalnych obrazów CBCT, podczas gdy SCT2 jest generowany z CBCT_cor.
ocena
W tym badaniu pacjentów z grupy walidacyjnej wykorzystano do oceny poprawy jakości obrazu. Średni błąd bezwzględny (MAE) i średni błąd (ME) obliczono odpowiednio dla CBCT, CBCT_cor, SCT1 i SCT2 względem RCT w obszarze masek binarnych. W międzyczasie porównano również profile HU dla tego typu obrazów, przeprowadzając porównanie side-by-side.
Plany terapii łukiem zmodyfikowanym wolumetrycznie (VMAT) dla pacjentów w grupie walidacyjnej zostały utworzone na podstawie obrazów RCT. Przepisana dawka wynosiła 69,96 Gy, 60,06 Gy i 56,1 Gy w celu wykreślenia docelowej objętości guza pierwotnego nosogardzieli, obustronnych końcowych węzłów chłonnych (PTV6996), regionu wysokiego ryzyka (PTV6006), regionu niskiego ryzyka i obustronnych obszarów węzłowych niskiego ryzyka (PTV5610) odpowiednio w 33 frakcjach. Punkty orientacyjne zostały przepisane z obrazów RCT na obrazy CBCT, CBCT_cor, SCT1 i SCT2 poprzez ścisłą rejestrację. Obliczenia dawki przeprowadzono przy użyciu Pinnacle3 (Wersja 16.2, Philips Radiation Oncology Systems, Madison, WI).
Porównanie dystrybucji dawki przeprowadzono między obrazami CBCT_cor, RCT, SCT1 i SCT2. Zebrano kilka parametrów dozymetrycznych do porównań ilościowych. Dla PTV, D2 (dawka odpowiadająca 2% objętości) dmieć na myśli (średnia dawka) i dr98 (dawka odpowiadająca 98% objętości). Lamjadaw, zmmieć na myśli lub drpowyższe (maksymalna dawka) została porównana. Globalne wskaźniki gamma pass 3D zostały również obliczone przez moduł radioterapii 3D-Slicer z parametrami odpowiednio 3%/3mm i 2%/2mm, z dawką graniczną 10%.
Test klasyfikacji miejsc Wilcoxona (pomiędzy SCT2 i CBCT, SCT2 i CBCT_cor, SCT2 i SCT1) przeprowadzono dla MAE, ME, współczynnika przepuszczania promieniowania gamma i parametrów dozymetrycznych opisanych wcześniej. Do wykonania tych testów wykorzystano Statistical Package for the Social Sciences (SPSS 21.0; SPSS Inc., Chicago, IL, USA). S<0,05 uznano za istotne statystycznie.

„Nieuleczalny myśliciel. Miłośnik jedzenia. Subtelnie czarujący badacz alkoholu. Zwolennik popkultury”.






More Stories
Emdoor przygotowuje się do zaprezentowania swoich osiągnięć w zakresie nowej technologii sztucznej inteligencji podczas targów Global Sources Mobile Electronics Show 2024.
LinkedIn wykorzystuje Twoje dane do szkolenia Microsoft, OpenAI i jego modeli AI – oto jak to wyłączyć
Zapomnij o Apple Watch Series 10 — Apple Watch Ultra 2 w kolorze Satin Black to smartwatch, który warto mieć